Howtos cluster i-Trop
| Description | HowTos pour le cluster i-Trop |
|---|---|
| Auteur | Ndomassi TANDO (ndomassi.tando@ird.fr) |
| Date de création | 08/11/19 |
| Date de modification | 01/10/25 |
Sommaire
- Preambule: Architecture du cluster i-Trop et logiciel à installer
- How to: Ajouter son couple de clés ssh pour se connecter au cluster
- How to: Transfert de fichiers via le sftp de filezilla sur le cluster
- How to: Se connecter au cluster via
ssh - How to: Réserver un ou plusieurs coeurs sur un noeud de calcul
- How to: Transférer mes fichiers depuis le serveur nas vers les noeuds
- How to: Utiliser les Module Environment
- How to: Lancer un job avec Slurm
- How to: Choisir une partition
- How to: Voir ou supprimer vos données sur les /scratch des noeuds
- How to: Utiliser un conteneur singularity
- How to: Citer le plateau dans vos publications
- Links
- License
Préambule
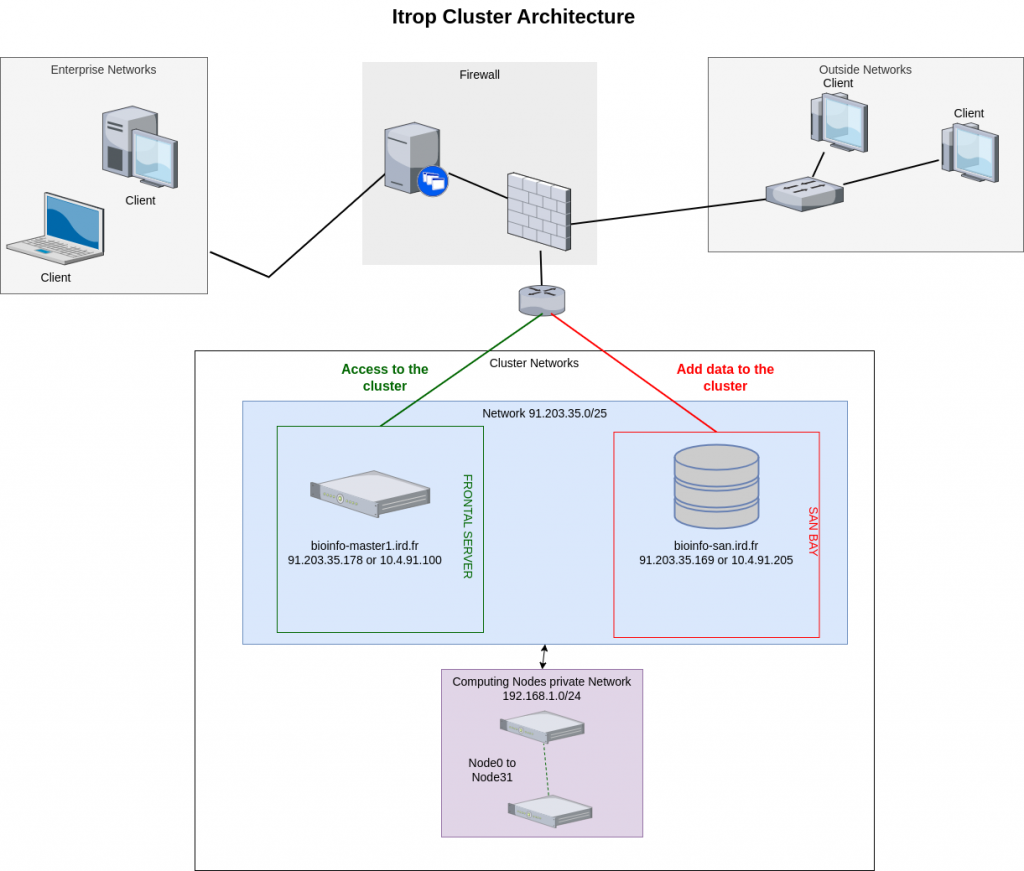
Architecture du cluster i-Trop :
Le cluster de calcul i-Trop est composé d’un ensemble de serveurs de calcul accessibles via une machine frontale. Les connexions à ces serveurs de calculs se font grâce à cette machine maître qui assure la répartition des différentes analyses entre les machines disponibles à un moment donné.
Le cluster de calcul est composé de :
- 1 machine maître
- 1 baie san permettant un stockage temporaire de données projet jusqu’à 800To
- 32 noeuds de calcul CPU d’une capacité totale de 1148 coeurs et 6329Go de RAM et un serveur GPU de 8 cartes graphiques RTX 2080.
En voici l’ architecture:
Se connecter à un serveur en ssh depuis une machine Windows
| Système | Logiciel | Description | url |
|---|---|---|---|
 |
mobaXterm | un terminal avancé pour Windows avec un serveur X11 et un client SSH | Télécharger |
 |
putty | Putty permet de se connecter à un serveur Linux depuis une machine Windows . | Télécharger |
Transférer des fichiers depuis son ordinateur vers des serveurs Linux avec SFTP
| Systèmes | logiciel | Description | url |
|---|---|---|---|
   |
 filezilla filezilla |
client FTP et SFTP | Télécharger |
Voir et éditer ses fichiers en local ou sur un serveur distant
| Type | Logiciel | url |
|---|---|---|
| Distant, console mode | nano | Tutorial |
| Distant, console mode | vi | Tutorial |
| Distant, graphic mode | komodo edit | Télécharger |
| Linux & windows éditeur | Notepad++ | Télécharger |
How to : Ajouter son couple de clés ssh pour se connecter au cluster
Suivre les instructions ici: https://bioinfo.ird.fr/index.php/en/tutorials-howtos-add-ssh-keys/
How to : Transfert de fichiers via le sftp de filezilla sur le cluster
Télécharger et installer FileZilla
Ouvrir FileZilla et sauvegarder les accès au cluster i-Trop dans le manager de site
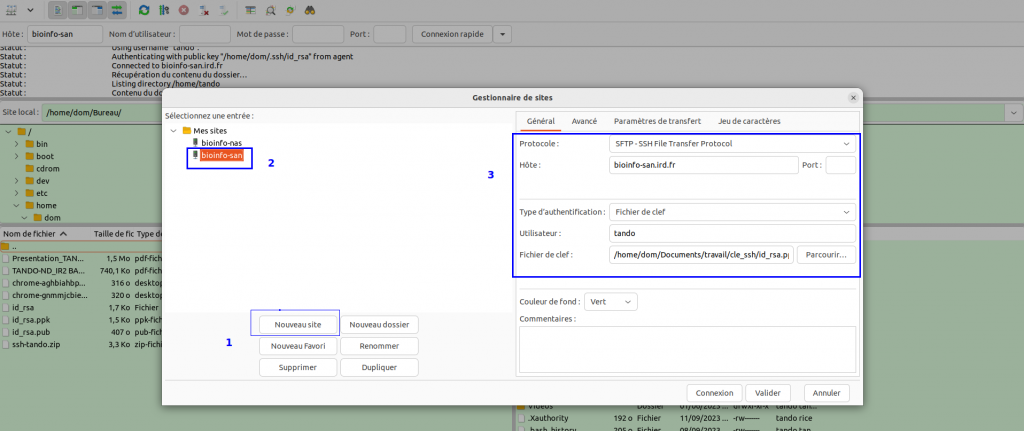
Dans le menu de FileZilla, aller dans File > Site Manager. Puis suivre ces 5 étapes:
- Cliquer sur New Site.
- Ajouter un nom explicite pour ce site.
- Renseigner l’hôte bioinfo-san.ird.fr
- Mettre le Logon Type à « Normal » et taper vos identifiants de connexion au cluster
- Choisir le port 22 et appuyer sur le bouton « Connect ».
Transfèrer des fichiers
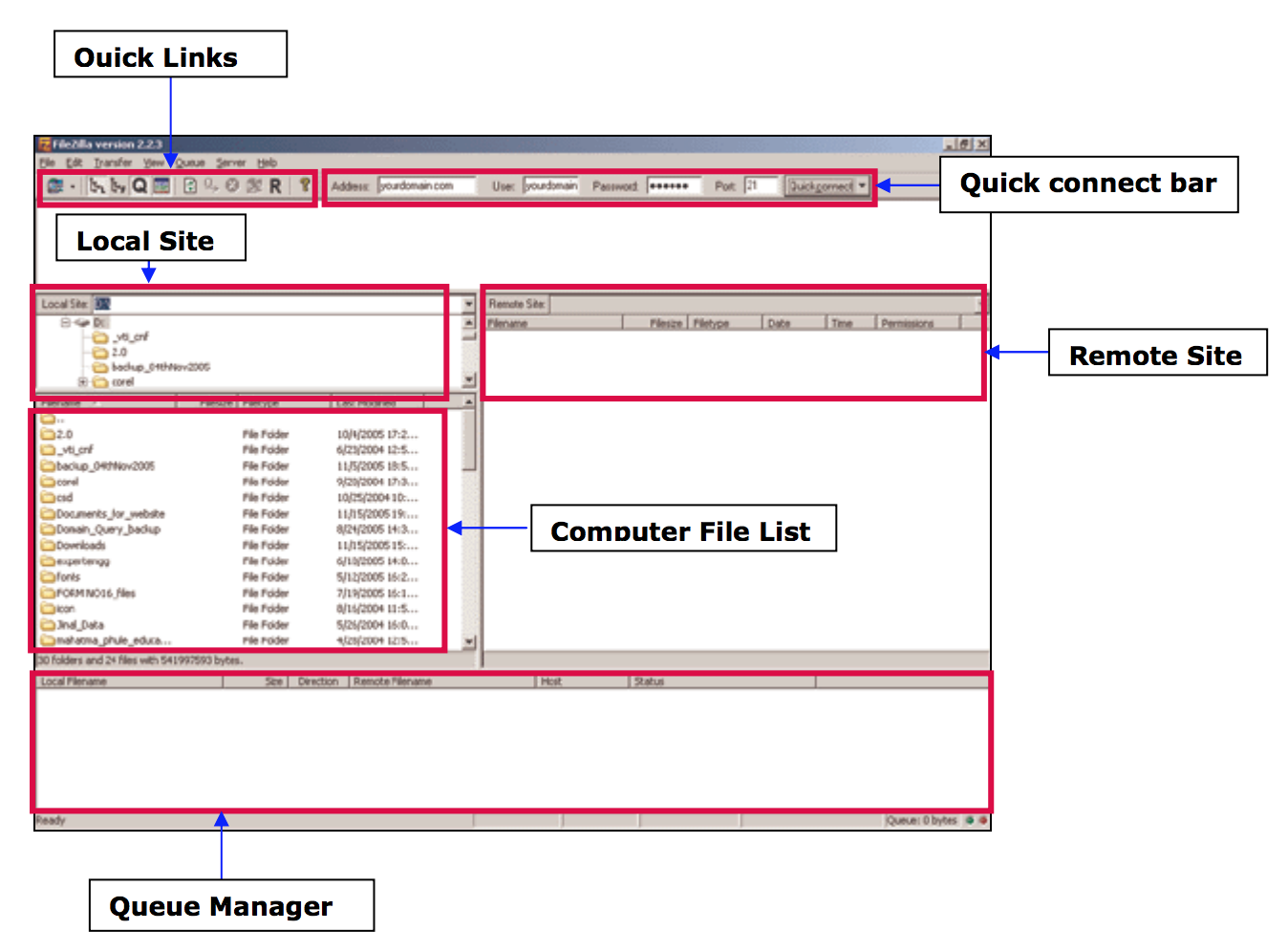
- Depuis votre ordinateur vers le cluster : faire un glisser-déposer d’un document depuis la colonne de gauche vers la colonne de droite.
- Depuis le cluster vers votre ordinateur : aire un glisser-déposer d’un document depuis la colonne de droite vers la colonne de gauche
How to : Se connecter au cluster via ssh
Depuis un ordinateur Windows :
Avec mobaXterm:
- Cliquer sur le bouton session et cliquer sur SSH.
- Dans la case remote host, taper: bioinfo-master1.ird.fr
- Cocher la case specify username et entrer votre login
- Choisir Advanced SSH settings
- Cocher la case « Use private key » et fournir le fichier de clé id_rsa.ppk de votre ssh-login.zip récupéré plus tôt et taper OK
Depuis un ordinateur MAC ou Linux:
Ouvrir l’application terminal et taper la commande suivante:
ssh login@bioinfo-master1.ird.fr
avec login: votre compte cluster
How to : Réserver un ou plusieurs coeurs en mode intéractif
Le cluster utilise Slurm (https://slurm.schedmd.com/documentation.html) pour gérer les analyses des utilisateurs
Il surveille les ressources disponibles (CPU et RAM ) et les alloue aux utilisateurs pour lancer leurs jobs.
En étant connectés au cluster, vous avez la possibilité de réserver un ou plusieurs coeurs des 27 noeuds de calcul disponibles.
Réserver un coeur
Taper la commande suivante:
srun -p short --pty bash -i
Vous serez connecté de manière aléatoire sur un des noeuds de la partition short avec un coeur réservé.
Réserver plusieurs coeurs à la fois
Taper la commande suivante:
srun -p short -c X --pty bash -i
Avec X le nombre de coeurs choisis entre 2 et 12.
Vous serez connecté de manière aléatoire sur un des noeuds de la partition short avec X coeurs réservés
Réserver un coeur sur un noeud spécifique:
Taper la commande suivante
srun -p short --nodelist=nodeX --pty bash -i
Avec nodeX qui appartient à la partition short
Réserver une caractéristique d’un noeud avec l’option –constraint:
Les Features/Constraints sur un noeud permettent aux utilisateurs de faire des requêtes spécifiques à Slurm. Sur notre cluster, nous avons plusieurs « features » qui dépendent de ce que vous souhaitez:
| Features | Description | Noeuds |
|---|---|---|
| 512 | Permet de sélectionner des noeuds de 512Go dans la partition highmem | node5, node28,node29,node30 |
| avx | Permet de sélectionner des noeuds avec des processeurs avx | node0, node1, node4, node5, node7, node17, node18, node20, node21, node22, node23, node24, node25, node26, node27, node28, node29, node31 |
| BEAST | Permet de sélectionner des noeuds pouvant utiliser BEAST avec beagle | node4, node7, node28, node29, node30 |
| dell | Permet de sélectionner des noeuds de la marque Dell | node28,node29,node30 |
| geforce | Permet de sélectionner des noeuds avec une carte graphique geforce | node26 |
| infiniband | Permet de sélectionner des noeuds avec de l’infiniband pour accélérer vos transferts de données | node0, node1, node4, node5, node7, node8, node11, node17, node20, node21, node22, node24, node25, node26, node27 |
| rtx2080 | Permet de sélectionner des noeuds gpu avec des cartes rtx2080 | node26 |
| xeon | Permet de sélectionner des noeuds avec des processeurs intel xeon | node0, node1, node5,node8, node10, node11, node12, node13, node14, node15, node16, node17, node18, node20, node21, node22, node23, node24, node25, node27 |
| xeon-gold | Permet de sélectionner des noeuds avec des processeurs intel xeon-gold | node4, node7, node26, node28, node29, node30, node31 |
Pour utiliser une ou plusieurs de ces features, vous devez utiliser l’option --constraint
Pour exemple, pour choisir un noeud avec des processeurs avx, vous taperez:
srun --constraint=avx --pty bash -i
Par exemple, pour choisir un noeud avec 512Go de RAM et en infiniband sur la partition highmem, vous taperez:
srun -p highmem --constraint=512,infiniband --pty bash -i
How to : Transférer mes données depuis le serveur san vers les noeuds
Sur le cluster, chaque noeud a sa propre partition appelée /scratch.
/scratch est utilisée pour recevoir temporairement les données à analyser, effectuer les analyses et accueillir les résultats
Les données sur les partitions /scratch sont conservées au maximum 30 jours sauf sur les noeuds de la partition long jusqu’à 45 jours
Il est obligatoire de transférer ses données vers le /scratch du noeud réservé avant de lancer ses analyses.
Les volumes /scratch volume vont de 1To à 14To en fonction du noeud choisi.
Quand les analyses sont finies, pensez à récupérer vos données.
La commande scp :
Pour transférer des données entre 2 serveurs distants, nous utilisons la commande scp
scp -r source destination
Il existe 2 syntaxes possibles:
Récupérer des données depuis un serveur distant:
scp -r remote_server_name:path_to_files/file local_destination
Transférer des données vers un serveur distant:
scp -r /local_path_to_files/file remote_server_name:remote_destination
Description des partitions:
Données utilisateurs
Chaque utilisateur a un volume de 100Go pour héberger des données
Ce répertoire personnel est hébergé dans la partition /users de bioinfo-san.ird.fr
Par exemple pour l’utilisateur test, son répertoire personnel sera /users/test
Attention: Sur bioinfo-master1.ird.fr et les noeuds de calcul, les répertoires /users/ et /home sont équivalents. Ce qui n’est pas le cas sur bioinfo-master.ird.fr ou /users et /home ne sont pas les mêmes
Les partitions projets
- /projects/medium: héberge les projets petits ou temporaires jusqu’à 1To
- /projects/large: héberge des des projets classiques de 1 à 4,9To
- /projects/xl:héberge des gros projets de 5 à 9,9To
La partition share
la partition /share est utilisée pour héberger des banques de données ou rendre disponible des données temporaires.
Utiliser les noeuds infiniband pour des transferts plus rapides:
srun -p highmem --constraint=infiniband --pty bash -iVous êtes désormais sur un noeud qui a accès au réseau infiniband, pour l’utiliser, modifiez juste l’alias san en san-ib pour faire vos transfertsy
Il y a 2 syntaxes possibles:
Récupérer des données depuis le san:
scp -r san-ib:path_to_files/file local_destination
Envoyer des données vers le san:
scp -r /local_path_to_files/file san-ib:remote_destination
How to : Utiliser les module Environnement
Module Environment vous permet de changer dynamiquement vos variables d’environnement(PATH, LD_LIBRARY_PATH) et choisir vos versions de logiciels.
La syntaxe utilisée pour définir ces modules est /.
Les logiciels sont divisés en 2 groupes:
- bioinfo: liste tous les logiciels de bioinformatiques
- system: liste tous les logiciels systèmes tels que python, perl etc…
Afficher les logiciels disponibles
module avail
Afficher la descripton d’un logiciel
module whatis module_name/version
avec module_name: le nom du module.
Par exemple : Pour la version 1.7 de samtools:
module whatis samtools/1.7
Charger une version de logiciel:
module load module_name/version
avec module_name: le nom du module.
Par exemple : Pour la version 1.7 de samtools:
module load samtools/1.7
Décharger un logiciel
module unload module_name/version
avec module_name: le nom du module.
Par exemple : pour la version 1.7 de samtools:
module unload samtools/1.7
Afficher tous les modules chargés
module list
Décharger tous les modules
module purge
How to : Lancer a job avec Slurm
Le cluster utilise l’ordonnaceur Slurm pour gérer et prioriser les jobs des utilisateurs.
Celui-ci contrôle les ressources disponibles (CPU et RAM) et les allouer en fonction des besoins utilisateurs pour leurs analyses.
Connecté à bioinfo-master1.ird.fr, on peut lancer une commande avec la commande srun ou un script en utilisant la commande sbatch.
Utiliser srun avec un commande:
La commande suivante permet d’allouer les ressources Slurm (noeuds, mémoire, coeurs) et de lancer la commande passée en arguments.
$ srun + commandExemple:
$ srun hostnamePermet d’obtenir le nom du noeud sur lequel la ressource est reservée.
Use sbatch to launch a script:
Le mode batch permet de lancer une analyse en suivant les étapes définies dans un script.
Slurm permet d’utiliser différents langages de scripts tels que bash, perl ou python.
Slurm alloue ensuite les ressources désirées et lance les analyses sur ces ressources en arrière plan.
Pour être interprété par Slurm, un script doit contenir une entête contenant toutes les options Slurm précédées par le nom clé #BATCH.
Exemple de script Slum:
#!/bin/bash
## Définir le nom du job
#SBATCH --job-name=test
## Définir le fichier de sortie
#SBATCH --output=res.txt
## Définir le nombre de tâches
#SBATCH --ntasks=1
## Définir la limite d'exécution
#SBATCH --time=10:00
## Définir 100Mo de mémoire par cpu
#SBATCH --mem-per-cpu=100
sleep 180 #lance une pause de 180sPour lancer une analyse à patir d’un script script.sh:
$ sbatch script.shAvec script.sh le nom du script à utiliser
Plus d’options Slurm ici: Slurm options
exemples de script pour slurm :
How to: Choisir une partition
Selon le type de jobs(analyses) que vous souhaitez lancer, vous avez le choix entre différentes partitions.
Les partitions sont des files d’attentes d’analyses avec chacune des priorités et des contraintes spécifiques telles que la taille ou le temps limite d’un job, les utilisateurs autorisés à l’utiliser etc…
Les jobs sont classés par priorité et traités grâce aux ressources (CPU et RAM) des l noeuds constituant ces partitions.
| partition | role | liste des noeuds | Nombre de coeurs | Ram |
|---|---|---|---|---|
| short | Jobs courts < 1 jour (priorité haute,jobs intéractifs) | node13,node14,node15,node16,node18,node27 | 12 à 24 coeurs | 48 à144Go |
| normal | jobs d’une durée de 7 jours maximum | node0,node1,node3,node4,node7,node9,node13,node17,node20, node21,node22,node23,node24,node27,node30 | 12 à 112 coeurs | 64 à 512Go |
| long | <7 jours< long jobs< 45 jours | node8,node10,node12 | 12 à 24 coeurs | 48 Go |
| highmem | jobs avec des besoins mémoire | node25,node28,node29 | 24 à 112 coeurs | 512 Go et 1To |
| supermem | jobs avec des besoins mémoire important | node2 | 256 coeurs | 2 To |
| gpu | Besoins d’analyses sur des coeurs GPU | node26 | 24 cpus et 8 GPUS coeurs | 192 Go |
A noter que l’accès à la partition gpu est restreint, une demande peut être faite ici: request access to gpu
La partition peut âtre choisie en suivant ce schéma:
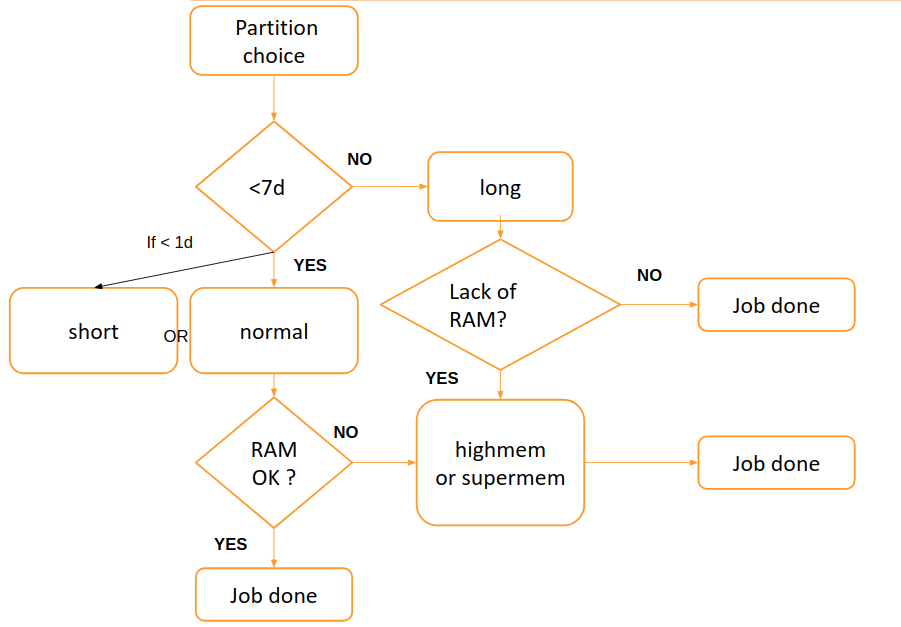
Par défaut, la partition choisie est la partition normal
Attention, highmem doit seulement être utilisé pour les jobs nécessitant au moins 35-40Go de mémoire.
La partition supermem partition doit être utilisé pour les assemblages volumineux et les jobs nécessitant plus de 100Go de mémoire
Vous pouvez utiliser la commande htop sur un noeud pour visualiser la mémoire consommée par un processus.
Pour choisir une partition, utiliser l’option -p.
sbatch -p partitionsrun -p partition
Avec partition, la partition choisie.
How to : Voir et supprimer vos données contenue dans la partition /scratch des noeuds
Les 2 scripts sont dans /opt/scripts/scratch-scripts/
- Pour voir vos données contenues dans les /scratchs des noeuds:
sh /opt/scripts/scratch-scripts/scratch_use.shet suivre les instructions - Pour supprimer vos données contenues dans les /scratchs des noeuds:
sh /opt/scripts/scratch-scripts/clean_scratch.shet suivre les instructions
How to : Utiliser un conteneur singularity
Singularity permet aux utilisateurs d’avoir un contrôle total de leur environnement . Les conteneurs Singularity peuvent être utilisés pour embarquer des workflows entiers, des logiciels, des librairies et même des données.
Singularity est installé sur le cluster en plusieurs versions
les conteneurs sont dans /usr/local/bioinfo/containers
Il faut charger en premier l’environnement avec:
module load singularity ou module load singularity/version_souhaitee
Obtenir de l’aide:
Utiliser la commande:
singularity help /usr/local/bioinfo/containers/singularity_version/container.simg
avec container.simg le nom du conteneur
avec singularity_version: la version voulue
Connexion shell au conteneur:
singularity shell /usr/local/bioinfo/containers/singularity_version/container.simg
Lancer un conteneur avec une seule application:
singularity run /usr/local/bioinfo/containers/singularity_version/container.simg + arguments
Lancer un conteneur qui a plusieurs applications:
singularity exec /usr/local/bioinfo/containers/singularity_version/container.simg + tools + arguments
Rajouter l’accès à un répertoire de l’hôte depuis le conteneur.
Utiliser l’option option --bind /host_partition:/container_partition
Exemple:
singularity exec --bind /toto2:/tmp /usr/local/bioinfo/containers/singularity_version/container.simg + tools + arguments
Le conteneur accèdera aux fichier de la partition /toto2 de la machine hôte dans sa partition /tmp
Par default, les partitions /home, /users, /opt,/scratch, /projects/medium, /projects/large, projects/xl et /share sont déjà montées.
How to : Citer le plateau i-Trop dans vos publications
Merci de copier la phrase suivante :
The authors acknowledge the ISO 9001 certified IRD i-Trop HPC (South Green Platform) at IRD montpellier for providing HPC resources that have contributed to the research results reported within this paper.
URL: https://bioinfo.ird.fr/- http://www.southgreen.fr
Links
- Cours liés : Linux for Dummies
- Cours liés : HPC
- Tutorials : Linux Command-Line Cheat Sheet
License
 i
i





